Investigadores del CSIC han aportado luz al mecanismo molecular por el que las bacterias intercambian información. Para ello han recreado un modelo tridimensional de la unión entre dos proteínas, RapF y PhrF, que regulan este sistema de señalización. Entender cómo se produce esta interacción podría abrir las puertas a nuevas aplicaciones en biotecnología
Las bacterias se comunican entre sí para coordinar procesos esenciales como la reproducción y la migración celular. Una investigación del Consejo Superior de Investigaciones Científicas (CSIC) ha desvelado detalles del mecanismo con el que estos microorganismos intercambian información.
El hallazgo fue recogido en el último número de la revista PLOS Biology y se basa en la recreación tridimensional de la estructura de la proteína RapF.
Los mecanismos biológicos de comunicación entre las bacterias se basan, principalmente, en sistemas de señalización de dos componentes: un sensor y un regulador de respuesta.
Alberto Marina, investigador del CSIC en el Instituto de Biomedicina de Valencia y responsable de la investigación, explica: “Además de estos dos componentes, habíamos observado que unas proteínas auxiliares llamadas conectores también participan en este complejo proceso de señalización”.
Estos conectores, denominados Rap, se asocian a proteínas Phr para modular la actividad de los reguladores de respuesta.
La reconstrucción en 3D de la proteína RapF junto a su péptido asociado PhrF ha permitido identificar un conjunto de aminoácidos responsables de la unión y de su especificidad funcional.
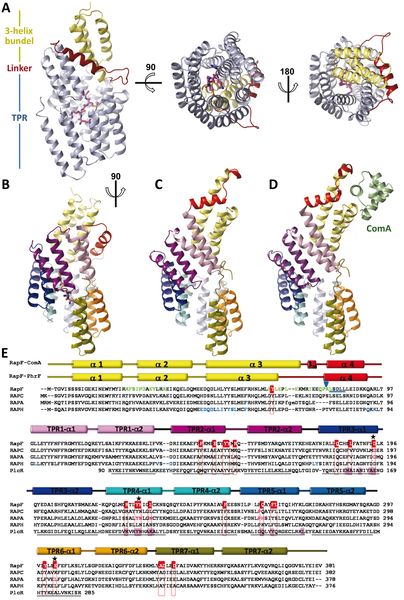
La investigadora del CSIC y corresponsable del trabajo, Francisca Gallego, precisa: “Experimentos posteriores nos permitieron relajar la especificidad de la unión RapF-PhrF simplemente cambiando uno de esos aminoácidos, lo que equivale a decir que hemos podido modificar su función”.
Para Marina, “el descubrimiento de estos residuos y su sistema de funcionamiento constituye un hallazgo clave para entender los mecanismos de función e inhibición de las proteínas Rap y abre la posibilidad de rediseñar artificialmente este tipo de proteínas de modo que sirvan para reprogramar vías de señalización biológicas, lo que podría tener múltiples aplicaciones en el campo de la biotecnología”.
La investigación también ha contado con la participación del Centro de Investigación Biomédica en Red de Enfermedades Raras.
Francisca Gallego del Sol, Alberto Marina. «Structural Basis of Rap Phosphatase Inhibition by Phr Peptides» PLOS Biology. DOI:10.1371/journal.pbio.1001511
Fuente: Sinc. Aportado por Eduardo J. Carletti
Más información:
- Descubren cómo la bacteria "Bacillus subtilis" libera al medio copias de su genoma
- Mecanismo bacteriano mantiene sincronizado un grupo de robots
- Curiosidad: Una forma única de vida mitad planta, mitad animal, con un mecanismo único
- Con selección artificial crean una bacteria con un ADN modificado, sin timina
- Utilizando bacterias con funciones lógicas diseñan circuitos vivos
- Una nueva bacteria de vida extrema se come el casco del Titanic
- Organismos se protegen a sí mismos y su entorno convirtiéndolo en oro
- Científicos descubren transferencia electrónica interespecies en microorganismos
- Las bacterias más simples son capaces de oler
- ¿Se convirtieron las bacterias en células complejas mucho antes en la evolución de lo que se pensaba?